Ácido nucleico bloqueado
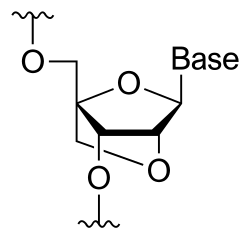
Un ácido nucleico bloqueado (en inglés, LNA, de locked nucleic acid), tamén coñecido como ácido nucleico ponteado (BNA, do inglés bridged nucleic acid) ou ARN inaccesible, é un ARN que presenta un nucleótido modificado. Mostran unha grande afinidade e especificidade con dianas de ADN nativas. O residuo de ribosa nun nucleótido de ácido nucleico bloqueado está modificado ao presentar unha ponte extra que conecta o oxíxeno 2' co carbono 4'. A ponte bloquea a conformación da ribosa nunha configuración 3'-endo (norte), a cal a miúdo se encontra en dúplex da forma A. Os nucleótidos de ácido nucleico bloqueado poden ser mesturados con residuos de ADN ou ARN dun oligonucleótido sempre que se desexe e despois hibridar este cun ADN ou ARN seguindo as regras de emparellamento de bases de Watson e Crick. Algúns destes oligómeros son sintetizados quimicamente e están dispoñibles comercialmente. A conformación da ribosa bloqueada mellora a colocación das bases unhas sobre outras (stacking) e a preorganización do esqueleto de azucre fosfato do ácido nucleico. Isto aumenta significativamente as propiedades de hibridación do oligonucleótido tales como a temperatura de desnaturalización.[1]
Os ácidos nucleicos bloqueados foron sintetizados de forma independente polo grupo de Jesper Wengel[2] en 1998, sendo sintetizados por primeira vez polo grupo de Takeshi Imanishi[3] en 1997. Os dereitos intelectuais sobre os ácidos nucleicos bloqueados son propiedade desde 1997 da compañía de biotecnoloxía danesa Exigon A/S.[4]

Os nucleótidos de ácido nucleico bloqueado utilízanse para incrementar a sensibilidade e a especificidade de expresión nas micromatrices de ADN, sondas FISH fluorescentes para técnicas de hibridación in situ, sondas para PCR en tempo real e outras técnicas de bioloxía molecular baseadas en oligonucleótidos. Polo momento (2005) a única técnica eficaz para a detección in situ de micro ARN é o uso de ácidos nucleicos bloqueados. Un triplete de nucleótidos de ácido nucleico bloqueado que rodea un só sitio con emparellamento de bases incorrecto maximiza a especificidade dunha sonda de ácido nucleico bloqueado a menos que a sonda conteña a base guanina do emparellamento G-T incorrecto.[5]
A utilización de oligonucleótidos baseados en ácidos nucleicos bloqueados como ferramentas terapéuticas é un campo emerxente en biotecnoloxía. A compañía farmacéutica danesa Santaris Pharma a/s é propietaria de todos os dereitos para a utilización terapéutica da tecnoloxía dos ácidos nucleicos bloqueados,[6] e actualmente está desenvolvendo un novo fármaco para o tratamento da hepatite C baseado nos ácidos nucleicos bloqueados, chamado Miravirsen, que ten como diana o xene miR-122, e a finais de 2010 este fármaco estaba en ensaio clínico en fase II.[7]
Utilidade da tecnoloxía dos ácidos nucleicos bloqueados
Algunhas das utilidades da súa utilización son:
- Son ideais para a detección de secuencias curtas de ADN e ARN.
- Incrementa a estabilidade térmica dos ácidos nucleicos bicatenarios.
- Teñen a capacidade de discriminar entre ácidos nucleicos que se diferencian nun só nucleótido.
- Son resistentes ás exo e endonucleases, o que lles dá unha alta estabilidade en aplicacións in vivo e in vitro.
- Alta especificidade no recoñecemento de secuencias diana.
- Facilitan a normalización da temperatura de fusión do ácido nucleico (Tm).
- A capacidade de invadir a febra do ácido nucleico permítelles detectar mesmo mostras con secuencias de difícil acceso.
- Son compatibles cos procesos encimáticos estandarizados.[8]
Aplicacións da tecnoloxía de ácidos nucleicos bloqueados
Algunhas das súas aplicacións son:
- PCR específica de alelos: permite o deseño de secuencias iniciadores máis curtas, sen comprometer a capacidade de unión ao sitio específico.[9]
- Permite xerar perfís de expresión xenética en micromatrices, aumentando a sensibilidade e a selectividade con cantidades máis pequenas de substrato.[10]
- Investigación do ARN pequeno.
- Xenotipificación de SNPs .
- Produción de oligonucleótidos de ARNm antisentido.
- Interferencia de ARN (ARNi).
- ADNcimas (DNAzymes).
- Sondas de polarización fluorescente.
- Balizas moleculares .
- Reparación de xenes e salto de exóns.
- Detección de variantes de empalme.
- Hibridación comparativa de xenomas (GCH).[11]
Existen outras aplicaciones terapéuticas ou diagnósticas que se encontran actualmente en desenvolvemento.[11]
Notas
- ↑ Kaur, H; Arora, A; Wengel, J; Maiti, S (2006). "Thermodynamic, Counterion, and Hydration Effects for the Incorporation of Locked Nucleic Acid Nucleotides into DNA Duplexes". Biochemistry 45 (23): 7347–55. PMID 16752924. doi:10.1021/bi060307w.
- ↑ Alexei A. Koshkin, Sanjay K. Singh, Poul Nielsen, Vivek K. Rajwanshi, Ravindra Kumar, Michael Meldgaard, Carl Erik Olsen, Jesper Wengel (1998). "LNA (Locked Nucleic Acids): Synthesis of the adenine, cytosine, guanine, 5-methylcytosine, thymine and uracil bicyclonucleoside monomers, oligomerisation, and unprecedented nucleic acid recognition". Tetrahedron 54 (14): 3607–30. doi:10.1016/S0040-4020(98)00094-5.
- ↑ Satoshi Obika, Daishu Nanbu, Yoshiyuki Hari, Ken-ichiro Morio, Yasuko In, Toshimasa Ishida, Takeshi Imanishi (1997). "Synthesis of 2′-O,4′-C-methyleneuridine and -cytidine. Novel bicyclic nucleosides having a fixed C3'-endo sugar puckering". Tetrahedron Lett. 38 (50): 8735–8. doi:10.1016/S0040-4039(97)10322-7.
- ↑ "Copia arquivada". Arquivado dende o orixinal o 03 de xaneiro de 2011. Consultado o 27 de novembro de 2015. Arquivado 03 de xaneiro de 2011 en Wayback Machine.
- ↑ You Y., Moreira B.G.; Behlke M.A. and Owczarzy R. (2006). "Design of LNA probes that improve mismatch discrimination". Nucleic Acids Res. 34 (8): e60. PMC 1456327. PMID 16670427. doi:10.1093/nar/gkl175.
- ↑ "Developing LNA technology for new-generation cancer drugs" (PDF). SP2 Magazine. marzo.
- ↑ Franciscus, Alan (2010). "Hepatitis C Treatments in Current Development". HCV Advocate. Arquivado dende o orixinal o 15 de agosto de 2015. Consultado o 27 de novembro de 2015. Arquivado 15 de agosto de 2015 en Wayback Machine.
- ↑ "Locked Nucleic Acid Technology". Arquivado dende o orixinal o 20 de agosto de 2009. Consultado o 27 de novembro de 2015. Arquivado 20 de agosto de 2009 en Wayback Machine.
- ↑ Bonetta, Laura (2005). "Prime time for real-time PCR". Nat. Methods 2 (4): 305–312. doi:10.1038/nmeth0405-305.
- ↑ Roberts, Peter (2006). "MicroRNA expression profiling on arrays enhanced with locked nucleic acids". Nat. Methods 3 (4). doi:10.1038/nmeth869.
- ↑ 11,0 11,1 "Copia arquivada". Arquivado dende o orixinal o 01 de setembro de 2010. Consultado o 27 de novembro de 2015. Arquivado 01 de setembro de 2010 en Wayback Machine.
Véxase tamén
Ligazóns externas
- LNA Oligo Tools and Design Guidelines Arquivado 20 de agosto de 2009 en Wayback Machine.
- LNA Oligo melting temperature including mismatches
- LNA Summary
- Vester B, Wengel J (2004). "LNA (locked nucleic acid): high-affinity targeting of complementary RNA and DNA". Biochemistry 43 (42): 13233–41. PMID 15491130. doi:10.1021/bi0485732.
- Petersen M, Wengel J (2003). "LNA: a versatile tool for therapeutics and genomics". Trends Biotechnol. 21 (2): 74–81. PMID 12573856. doi:10.1016/S0167-7799(02)00038-0.
- Ng PS, Bergstrom DE (2005). "Alternative nucleic acid analogues for programmable assembly: hybridization of LNA to PNA". Nano Lett. 5 (1): 107–11. PMID 15792422. doi:10.1021/nl048246f.