作用機序
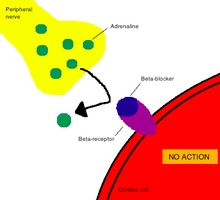
薬理学における作用機序(さようきじょ、英: mechanism of action, MOA)とは、薬剤がその薬理学的効果を発揮するための特異的な生化学的相互作用を意味する[2]。作用機序では大抵、薬剤が結合する酵素あるいは受容体といった特定の分子標的について言及される[3]。受容体部位は、薬物の化学構造とそこで起こる特定の作用に基づき、薬物に対する特定の親和性を持つ。
受容体に結合しない薬物は、単に体内の化学的または物理的特性と相互作用することにより、対応する治療効果を生じる。このように作用する薬物の一般的な例としては、制酸剤や下剤がある[2]。
対照的に、作用機構 (英: mode of action, MoA) は、生体が物質に曝露されることによって生じる細胞レベルでの機能的または解剖学的変化を意味する。
重要性
新しい薬物・医薬品の作用機序を解明することは、いくつかの理由から重要である。
- 抗感染症薬の開発では、臨床安全性に関わる問題を予測することができる。たとえば、細胞膜や電子伝達系を破壊する薬物は、ヒト細胞には存在しない細胞壁成分 (ペプチドグリカンやβグルカン) や70Sリボソームを標的とした薬物よりも毒性の問題を引き起こす可能性がある[4][5]。
- 薬剤の特定の部位と受容体の間の相互作用を知ることにより、他の薬剤によってこの相互作用を再現することができ、したがって同じ治療効果を生み出すことができる。実際、この方法は新薬の開発にも使われている。
- どの患者が治療に反応する可能性が高いかを特定するのに役立つ。乳がん治療薬トラスツズマブはHER2タンパク質を標的とすることが知られているため、たとえば、腫瘍にHER2分子が存在するかどうかをスクリーニングして、患者がトラスツズマブ治療の恩恵を受けるかどうかを判断することができる[6][7]。
- 標的経路に対する薬物の効果を患者内で監視できるため、より良い投与 (英語版) が可能になる。例えば、スタチンの投与量は、通常、患者の血中コレステロール値を測定することによって決定される[6]。
- 薬剤耐性が出現する可能性が低くなるような方法で薬剤を組み合わせる(併用療法)ことができる。抗感染薬や抗がん剤がどのような細胞構造に作用するかを知ることで、複数の標的を同時に阻害するカクテルを投与することが可能となり、それにより微生物や腫瘍のDNAにおける単一の変異が薬剤耐性や治療の失敗につながるリスクを低減する[4][8][9][10]。
- 薬剤の他の適応症を特定できる場合がある。たとえば、シルデナフィルがホスホジエステラーゼ-5 (PDE-5) (英語版) タンパク質を阻害するという発見により、PDE-5は肺高血圧症の肺で発現していることから、肺動脈性肺高血圧症の治療薬として再利用することが可能となった[11][12]。
決定方法
顕微鏡を用いた手法

生理活性化合物は、標的細胞の表現型変化を誘導、顕微鏡で観察できる変化を誘発し、化合物の作用機序を洞察することができる[13]。
抗菌剤では、標的細胞のスフェロプラストへの変換は、ペプチドグリカン合成が阻害されていることを示す可能性があり、標的細胞の線維化は、PBP3、FtsZ、またはDNA合成が阻害されていることを示す可能性がある。その他の抗菌剤誘発性変化としては、卵形細胞の形成、偽多細胞形態、局所的な腫脹、水疱成形、出血、およびペプチドグリカンの肥厚が挙げられる[4]。抗がん剤の場合、濾過胞形成は、化合物が形質膜を破壊している徴候を示す可能性がある[14]。
このアプローチの現在の制限は、手動でデータを生成して解釈するのに必要な時間であるが、自動顕微鏡と画像解析ソフトウェアの進歩により、この問題が解決される可能性がある[4][13]。
直接生化学的方法
直接生化学的方法には、薬物候補のようなタンパク質または低分子を標識して、全身にトレースする方法が含まれる[15]。これは、薬物のファーマコフォアを同定するために、薬物のアウトラインの基本的な表現など、対象の小さな標的に結合する標的タンパク質を見つけるための最も直接的なアプローチであることが証明されている。標識分子とタンパク質との間の物理的相互作用により、生化学的手法を用いて、毒性、有効性、および薬物の作用機序を決定することができる。
計算推論法
計算推論法は通常、主にコンピュータベースのパターン認識に基づいて低分子薬物のタンパク質標的を予測するために使用される[15]。ただし、この方法は、既存の薬物や新規開発の薬物の新たな標的を見つけるためにも使用することができる。薬物分子のファーマコフォアを同定することで、パターン認識のプロファイリング法を、新たな標的が同定された場所で実行することができる[15]。これにより、薬物の特定の機能性分子がタンパク質上の特定の領域と相互作用し、治療効果をもたらすことが知られているため、作用機序の可能性についての洞察が得られる。
オーミクスに基づく手法
オーミクスに基づく方法は、逆遺伝学およびゲノミクス、トランスクリプトミクス、およびプロテオミクスなどのオーミクス技術を使用して、目的の化合物の潜在的な標的を同定する[16]。逆遺伝学およびゲノミクスアプローチは、例えば、遺伝的摂動 (例: CRISPR-Cas9またはsiRNA) を化合物と組み合わせて使用して、ノックダウンまたはノックアウトが化合物の薬理学的効果を無効化する遺伝子を同定する。一方、化合物のトランスクリプトミクスおよびプロテオミクスプロファイルは、既知の標的を持つ化合物のプロファイルと比較するために使用できる。計算推論のおかげで、化合物の作用機序に関する仮説を立てることが可能となり、その仮説を後で検証することができる[16]。
作用機序が知られている薬物
作用機序が知られている多くの薬がある。一例はアスピリンである。
アスピリン
アスピリンの作用機序は、酵素シクロオキシゲナーゼの不可逆的阻害を含み、プロスタグランジンとトロンボキサンの産生を抑制することによって痛みと炎症が抑えられる[17]。この作用機序はアスピリンに特有のものであり、すべての非ステロイド性抗炎症薬 (NSAIDs) に対して一定ではない。むしろ、アスピリンはCOX-1を不可逆的に阻害する唯一のNSAIDである[18]。
作用機序が不明な薬物
いくつかの薬物では作用機序がまだ不明である。しかし、ある薬物の作用機序が不明であっても、依然として機能する。その薬物がどのように受容体と相互作用して治療効果を発揮するのか不明なだけである。
作用機構
一部の文献論文では、作用機序 (mechanism of action, MOA (英語版) )と作用機構 (mode of action, MoA (英語版) ) という用語が互換的に使用されている。典型的には、薬物が相互作用して医学的効果をもたらす方法を指している。しかし、実際には、作用機構とは、生体が物質に曝露されることによって生じる細胞レベルでの機能的または解剖学的変化を意味する。これは、作用機序とは異なり、阻害、活性化、アゴニズム、またはアンタゴニズムを介したか否かにかかわらず、薬物自体と酵素または受容体との間の相互作用およびその特定の形態に焦点を当てた、より具体的な用語である。さらに、作用機序という用語は、主に薬理学で使用される主要項であり、一方、作用機構は微生物学や生物学の特定の側面の分野でより頻繁に使用されている。
脚注
- ^ Ogrodowczyk, M.; Dettlaff, K.; Jelinska, A. (2016). “Beta-blockers: Current state of knowledge and perspectives”. Mini Reviews in Medicinal Chemistry 16 (1): 40–54. doi:10.2174/1389557515666151016125948. PMID 26471965.
- ^ a b Spratto, G.R.; Woods, A.L. (2010). Delmar Nurse's Drug Handbook. Cengage Learning. ISBN 978-1-4390-5616-5
- ^ Grant, R.L.; Combs, A.B.; Acosta, D. (2010) "Experimental Models for the Investigation of Toxicological Mechanisms". In McQueen, C.A. Comprehensive Toxicology (2nd ed.). Oxford: Elsevier. p. 204. ISBN 978-0-08-046884-6.
- ^ a b c d e Cushnie, T.P.; O’Driscoll, N.H.; Lamb, A.J. (2016). “Morphological and ultrastructural changes in bacterial cells as an indicator of antibacterial mechanism of action”. Cellular and Molecular Life Sciences 73 (23): 4471–4492. doi:10.1007/s00018-016-2302-2. hdl:10059/2129. PMID 27392605.
- ^ Chang, C.C.; Slavin, M.A.; Chen, S.C. (2017). “New developments and directions in the clinical application of the echinocandins”. Archives of Toxicology 91 (4): 1613–1621. doi:10.1007/s00204-016-1916-3. PMID 28180946.
- ^ a b No authors listed (2010). “Mechanism matters”. Nature Medicine 16 (4): 347. doi:10.1038/nm0410-347. PMID 20376007.
- ^ Joensuu, H. (2017). “Escalating and de-escalating treatment in HER2-positive early breast cancer”. Cancer Treatment Reviews 52: 1–11. doi:10.1016/j.ctrv.2016.11.002. PMID 27866067.
- ^ Cihlar, T.; Fordyce, M. (2016). “Current status and prospects of HIV treatment”. Current Opinion in Virology 18: 50–56. doi:10.1016/j.coviro.2016.03.004. PMID 27023283.
- ^ Antony, H.A.; Parija, S.C. (2016). “Antimalarial drug resistance: An overview”. Tropical Parasitology 6 (1): 30–41. doi:10.4103/2229-5070.175081. PMC 4778180. PMID 26998432.
- ^ Bozic, I.; Reiter, J.G.; Allen, B.; Antal, T.; Chatterjee, K.; Shah, P.; Moon, Y.S.; Yaqubie, A.; Kelly, N.; Le, D.T.; Lipson, E.J.; Chapman, P.B.; Diaz, L.A.; Vogelstein, B., Nowak, M.A. (2013). “Evolutionary dynamics of cancer in response to targeted combination therapy”. eLife 2: Article ID e00747. doi:10.7554/eLife.00747. PMC 3691570. PMID 23805382.
- ^ Tari, L.; Vo, N.; Liang, S.; Patel, J.; Baral, C.; Cai, J. (2012). “Identifying novel drug indications through automated reasoning”. PLOS ONE 7 (7): Article e40946. doi:10.1371/journal.pone.0040946. PMC 3402456. PMID 22911721.
- ^ Hayardeny, L. (2014). Why is it important to know the mode of action of drugs? (Conference presentation) (English). New Frontiers in Neuroscience and Methods of Transdisciplinary Education Workshop, Tel Aviv University, Israel: Tel Aviv University. 2017年3月18日閲覧。
- ^ a b Fetz, V.; Prochnow, H.; Brönstrup, M.; Sasse, F. (2016). “Target identification by image analysis”. Natural Product Reports 33 (5): 655–667. doi:10.1039/c5np00113g. hdl:10033/621283. PMID 26777141.
- ^ Dubovskii, P.V.; Vassilevski, A.A.; Kozlov, S.A.; Feofanov, A.V.; Grishin, E.V.; Efremov, R.G. (2015). “Latarcins: versatile spider venom peptides”. Cellular and Molecular Life Sciences 72 (23): 4501–4522. doi:10.1007/s00018-015-2016-x. PMID 26286896.
- ^ a b c Schenone, M.; Dančík, V.; Wagner, B.K.; Clemons, P.A. (2013). “Target identification and mechanism of action in chemical biology and drug discovery”. Nature Chemical Biology 9 (4): 232–240. doi:10.1038/nchembio.1199. ISSN 1552-4450. PMC 5543995. PMID 23508189.
- ^ a b Wecke, T.; Mascher, T. (2011). “Antibiotic research in the age of omics: from expression profiles to interspecies communication”. Journal of Antimicrobial Chemotherapy 66 (12): 2689–2704. doi:10.1093/jac/dkr373. PMID 21930574.
- ^ Vane, J. R.; Botting, R. M. (2003). “The mechanism of action of aspirin”. Thrombosis Res. 110 (5-6): 255-258. doi:10.1016/S0049-3848(03)00379-7. PMID 14592543.
- ^ Sharma, S.; Sharma, S. C. (1997). “An update on eicosanoids and inhibitors of cyclooxygenase enzyme systems”. Indian Journal of Experimental Biology 35 (10): 1025–1031. ISSN 0019-5189. PMID 9475035.