Análise de componentes principais

A Análise de Componentes Principais (ACP) ou Principal Component Analysis (PCA) é um procedimento matemático que utiliza uma transformação ortogonal (ortogonalização de vetores) para converter um conjunto de observações de variáveis possivelmente correlacionadas num conjunto de valores de variáveis linearmente não correlacionadas chamadas de componentes principais. O número de componentes principais é sempre menor ou igual ao número de variáveis originais. Os componentes principais são garantidamente independentes apenas se os dados forem normalmente distribuídos (conjuntamente). O PCA é sensível à escala relativa das variáveis originais. Dependendo da área de aplicação, o PCA é também conhecido como transformada de Karhunen-Loève (KLT) discreta, transformada de Hotelling ou decomposição ortogonal própria (POD).
O PCA foi inventado em 1901 por Karl Pearson.[1] Agora, é mais comumente usado como uma ferramenta de Análise Exploratória de Dados e para fazer modelos preditivos. PCA pode ser feito por decomposição em autovalores (Valores Próprios) de uma matriz covariância, geralmente depois de centralizar (e normalizar ou usar pontuações-Z) a matriz de dados para cada atributo.[2] Os resultados de PCA são geralmente discutidos em termos pontuações (scores) de componentes, também chamados de pontuações de fatores (os valores de variável transformados correspondem a um ponto de dado particular), e carregamentos (loadings), i.e., o peso pelo qual cada variável normalizada original deve ser multiplicada para se obter a pontuação de componente.[3]
O PCA é a mais simples das verdadeiras análises multivariadas por autovetores (Vetores Próprios). Com frequência, sua operação pode ser tomada como sendo reveladora da estrutura interna dos dados, de uma forma que melhor explica a variância nos dados. Se visualizarmos um conjunto de dados multivariados em um espaço de alta dimensão, com 1 eixo por variável, o PCA pode ser usado para fornecer uma visualização em dimensões mais baixas dos mesmos dados, uma verdadeira "sombra" do objeto original quando visto de seu ponto mais informativo. Isto é feito usando-se apenas os primeiros componentes principais, de forma que a dimensionalidade dos dados transformados é reduzida.
O PCA é fortemente ligado à análise fatorial (Factorial Analysis); de fato, alguns pacotes estatísticos propositadamente confluem as técnicas. A verdadeira análise de fatores faz suposições diferentes sobre a estrutura subjacente dos dados e encontra os autovetores de uma matriz levemente diferente.
Detalhes
O PCA é matematicamente definido[4] como uma transformação linear ortogonal que transforma os dados para um novo sistema de coordenadas de forma que a maior variância por qualquer projeção dos dados fica ao longo da primeira coordenada (o chamado primeiro componente), a segunda maior variância fica ao longo da segunda coordenada, e assim por diante.
Seja a matriz de dados, XT, com média empírica nula (i.e., a média empírica (amostral) da distribuição foi subtraída dos dados), onde cada uma das n linhas representa uma repetição diferente do experimento, e cada uma das m colunas dá um tipo particular de dado (e.g., os resultados de uma determinada sonda). (Note-se que XT é definida aqui e não X propriamente dito, e o que estamos chamando de XT é por vezes denotado por X.) A decomposição em valores singulares de X é X = WΣVT, onde a matriz m × m W é a matriz de autovetores da matriz de covariância XXT, a matriz Σ é m × n e é uma matriz diagonal retangular com números reais não negativos na diagonal, e a matriz n × n V é a matriz de autovetores de XTX. Assim, a transformação PCA que preserva a dimensionalidade (i.e., que dá o mesmo número de componentes principais do que o número de variáveis originais) é dada por:
V não é definida unicamente no caso usual de m < n − 1, mas Y vai, com frequência, ser definida unicamente. Como W (por definição da SVD de uma matriz real) é uma matriz ortogonal, e cada linha de YT é simplesmente uma rotação da linha correspondente de XT. A primeira coluna de YT é feita das "pontuações" dos casos relativamente ao componente "principal", a próxima coluna tem a pontuação relativamente ao segundo componente "principal", e assim por diante.
Se desejarmos uma representação de dimensionalidade reduzida, pode-se projetar X ao espaço reduzido definido apenas pelos primeiros L vetores singulares, WL:
- onde com a matriz identidade retangular .
A matriz W de vetores singulares de X 'e equivalentemente a matriz W de autovetores da matriz de covariâncias C = X XT,
Dado um conjunto de pontos no espaço euclidiano, o primeiro componente principal corresponde a uma linha que passa através da média multidimensional e minimiza a soma dos quadrados das distâncias dos pontos à linha. O segundo componente principal corresponde ao mesmo conceito, depois de subtrair-se toda a correlação com o primeiro componente principal dos pontos. Os valores singulares (em Σ) são as raízes quadradas dos autovalores da matriz XXT. Cada autovalor é proporcional à porção de "variância" (mais precisamente da soma dos quadrados das distâncias dos pontos à média multidimensional dos mesmos) que é correlacionada com cada autovetor. A soma de todos os autovalores é igual à soma dos quadrados dos pontos à média multidimensional dos mesmos. O PCA essencialmente rotaciona o conjunto de pontos em torno da média de forma a alinhá-los com os componentes principais. Isto move o máximo possível de variância (usando uma transformação ortogonal) a algumas das primeiras dimensões. Os valores nas dimensões restantes, portanto, tendem a serem pequenos e podem ser descartados com o mínimo de perda de informação. O PCA é comumente utilizado dessa maneira para redução de dimensionalidade. O PCA tem a distinção de ser a melhor transformação ortogonal para manter o subespaço que tem a maior "variância" (como definida ha pouco). No entanto, essa vantagem tem o preço de exigir mais recursos computacionais se comparado com, por exemplo, a transformada discreta de cosseno (quando esta também for aplicável). Técnicas de redução de dimensionalidade não linear tendem a ser ainda mais dispendiosas (computacionalmente) do que o PCA.
O PCA é sensível à escala das variáveis. Se tivermos apenas duas variáveis de variâncias amostrais iguais e positivamente correlacionadas, então o PCA irá consistir de uma rotação de 45°, e os "carregamentos" (ou loadings) para as duas variáveis relativos ao componente principal serão iguais. Mas se multiplicarmos todos os valores da primeira variável por 100, então o componente principal será quase igual a essa variável, com uma pequena contribuição da outra variável, ao passo que o segundo componente será quase que alinhado com a segunda variável original. Isso significa que, sempre que as diferentes variáveis têm unidades diferentes (como massa e temperatura), o PCA é de certa forma um método arbitrário de análise de dados. Por exemplo, resultados diferentes seriam obtidos se Farenheit fosse usado em vez de Celsius. Note-se que o artigo original de Pearson foi intitulado "On Lines and Planes of Closest Fit to Systems of Points in Space" – "in space" (no espaço) implica o espaço físico euclidiano, no qual tais ressalvas não ocorrem. Uma maneira de tornar o PCA menos arbitrário é usar as variáveis renormalizadas para variância unitária.
Discussão
Faz-se necessária a subtração do vetor médio das componentes principais, ou seja, "centralização na média", quando se pretende usar o PCA para garantir que os primeiros componentes principais descrevam a direção de máxima variância. Se a subtração da média não for feita, os primeiros componentes principais podem corresponder mais ou menos à média dos dados. Uma média de zero é necessária para encontrar a base que minimiza o erro médio quadrático da aproximação dos dados.[5]
Assumindo-se uma média empírica nula, ou seja, a média empírica da distribuição foi subtraída do conjunto de dados, o componente principal w1 de um conjunto de dados X pode ser definido como:
(Ver arg max para a notação.) Com os primeiros k − 1 componentes, o k-ésimo componente pode ser encontrado subtraíndo-se os primeiros componentes principais de X:
e substituindo-se isso como o novo conjunto de dados cujo componente principal é obtido em
O PCA é equivalente a funções ortogonais empíricas (EOF), um nome que é usado em meteorologia.
Uma rede neural autoencoder com uma camada linear escondida é similar ao PCA. À convergência, os vetores de peso dos K neurônios na camada escondida formarão uma base para o espaço formado pelos primeiros K componentes principais. Diferente do PCA, essa técnica não necessariamente produz vetores ortogonais.
O PCA é uma técnica fundamental em reconhecimento de padrões. No entanto, não é otimizado para separabilidade de classes.[6] Uma alternativa é a LDA, que leva esse aspecto em consideração.
Propriedades e limitações do PCA
Como visto acima, os resultados do PCA dependem da escala das variáveis.
A aplicabilidade do PCA é limitada por certas premissas[7] feitas em sua derivação.
Calculando o PCA através do método da covariância
O cálculo do PCA usando o método da covariância é descrito nesta seção.[8] Note-se, porém, que é melhor usar a decomposição em valores singulares (com software padrão de álgebra linear).
O objetivo é transformar um dado conjunto de dados X de dimensão M num conjunto alternativo Y de dimensão menor L. Equivalentemente, deseja-se a matriz Y, onde Y é a Karhunen–Loève transform (KLT) da matriz X:
Organizar o conjunto de dados
Suponha-se um conjunto de dados sobre um conjunto de observações de M variáveis, onde o objetivo é reduzir os dados de forma que cada observação possa ser descrita com apenas L variáveis, L < M. Suponha-se, ainda, que os dados podem ser dispostos como um conjunto de N vetores de dados com cada representando uma única observação agrupada das M variáveis.
- Escreva como vetores coluna, cada um tendo M linhas.
- Disponha os vetores coluna em uma única matriz X de dimensões M × N.
Cálcular a média empírica
- Encontre a média empírica ao longo de cada dimensão m = 1, …, M.
- Disponha esses valores de média em um vetor de média empírica u de dimensões M × 1.
Calcule os desvios da média
A subtração de média é uma parte fundamental no cálculo de uma base de componentes principais que minimize o erro médio da aproximação dos dados.[9] Logo, centralizam-se os dados da seguinte forma:
- Subtraia o vetor de média empírica u de cada coluna da matriz de dados X.
- Armazene os dados centralizados na matriz M × N B.
- onde h é um vetor-linha 1 × N de todos 1s:
Encontre a matriz de covariância
- Encontre a matriz de covariância emírica M × M C do produto externo da matriz B consigo mesma:
-
- onde
- é o operador de valor esperado,
- é o operador de produto externo, e
- é o operador conjugado transposto. Note-se que se B consiste inteiramente de números reais, o que ocorre em diversas aplicações, a "conjugada transposta" é a mesma que a transposta usual.
- onde
Para casos onde B são matrizes simples o correto seria fazer B*.B e não o que esta listado acima, veja que se B é uma matriz de 300 linhas e 20 colunas ao fazermos sua transposta temos 20 linhas e 300 colunas e o produto B.B* se torna uma matriz 300 linhas por 300 colunas, logo não houve redução de dados e sim ampliação dos mesmos. Fazendo o inverso obtemos 20 linhas por 20 colunas como o esperado.
- Note-se que a informação nesta seção é um tanto imprecisa, já que produtos externos se aplicam a vetores. Para casos de tensors, produtos tensoriais deveriam ser usados, mas a matriz de covariância no PCA é uma soma de produtos externos entre os vetores de amostras; de fato, o mesmo poderia ser representado como B.B*.
Encontra-se os autovetores e autovalores da matriz de covariância
- Calcula-se a matriz V de autovetores que diagonaliza a matriz de covariância C:
- onde D é a matriz diagonal de autovalores de C. Esse passo tipicamente envolve o uso de um método numérico para o cálculo de autovetores e autovalores. Tais algoritmos são amplamente disponíveis como pacotes da maioria dos sistemas de álgebra matricial, como o Scilab, o R (linguagem de programação), Mathematica,[10] SciPy, GNU Octave, bem como VXL e OpenCV.
- A matriz D toma a forma de uma matriz diagonal M × M, onde
- é o m-ésimo autovalor da matriz de covariância C, e
- A matriz V, também M × M, contém M vetores coluna, cada um de tamanho M, que representa os M autovetores da matriz de covariância C.
- Os autovalores e autovetores são ordenados e casados. Cada m-ésimo autovalor corresponde ao m-ésimo autovetor.
- Reordenam-se as colunas da matriz de autovetor V e da matriz de autovalor D na ordem decrescente de autovalores.
Calcula-se a energia acumulativa para cada autovetor
- Os autovalores representam, de certa forma, a distribuição da energia nos dados ao longo de cada um dos autovetores, onde aqui os autovetores formam uma base para os dados. A energia acumulativa g para o m-ésimo autovetor é a soma do conteúdo de energia ao longo de todos os autovalores de 1 a m:
Seleciona-se um subconjunto de autovetores como vetores base
- Armazenam-se as primeiras L colunas de V como a matriz M × L W:
- onde
- Utiliza-se o vetor g como um guia na escolha de um valor adequado para L. O objetivo é escolher o menor valor possível para L que ao mesmo tempo garanta um valor suficientemente alto de g em termos percentuais. Por exemplo, pode-se escolher L de forma que a energia acumulativa g é acima de um certo limiar, e.g. 90%. Nesse caso, escolhe-se o menor valor de L tal que
Converte-se os dados originais para pontuações-Z
- Criar um vetor desvio padrão M × 1 s da raiz quadrada de cada elemento ao longo da diagonal da matriz de covariância C:
- Calcular a matriz M × N de pontuações-Z:
- (dividir elemento-por-elemento)
- Note-se que apesar desse passo ser útil em diversas aplicações (pois normaliza os dados em relação à variâncias), ele não é uma parte integral do PCA/KLT
Projetar as pontuações-Z dos dados na nova base
- Os vetores projetados são as colunas da matriz
- W* é a conjugada transposta da matriz de autovetores.
- As colunas da matriz Y representam as transformadas de Karhunen–Loeve (KLT) dos vetores de dados nas colunas da matriz X.
Software/código fonte
- No software livre R, as funções
princompeprcomppodem ser usadas para PCA;prcompusa a decomposição em valores singulares, que geralmente fornece uma melhor precisão numérica. Existe uma explosão recente em implementações de PCA em diversos pacotes do R, geralmente em pacotes de propósito específico. Veja: [1]. - Em Octave, o qual é um ambiente livre de programação compatível com o MATLAB, a função
princompcalcula a componente principal. - OpenCV
- Na Biblioteca NAG, implementa-se PCA com a rotina
g03aa(disponível tanto em Fortran[11] quanto na linguagem C[12]). - Cornell Spectrum Imager- Uma ferramenta de código aberto baseada no ImageJ. Permite análise rápida e fácil de PCA para 3D datacubes.
- Weka também calcula componentes principais (javadoc).
Cálculo eficiente de componentes principais
Algoritmos iterativos
Em implementações práticas, especialmente para dados de alta dimensão (m grande), o método de covariância não é muito usado por não ser eficiente. Uma maneira de calcular o primeiro componente principal eficientemente[13] é dado no pseudo-código a seguir, para uma matriz de dados XT com média zero, sem precisar calcular sua matriz de covariância. Note-se que, aqui, uma matriz de dados com média nula significa que as colunas de XT devem ter, cada uma, média zero.
um vetor aleatório faça c vezes: (um vetor de tamanho m) para cada linha retorne
Esse algoritmo é simplesmente uma maneira eficiente de calcular XXTp, normalizando, e colocando-se o resultado de volta em p (en:Power iteration). Ele evita as nm² operações de cálculo da matriz de covariância. p ficará tipicamente próximo à primeira componente principal de XT dentro de poucas iterações, c. (A magnitude de t será maior depois de cada iteração. A convergência será detectada quando aumenta muito pouco relativo à precisão da máquina.)
Componentes principais subsequentes podem ser calculados subtraindo-se o componente p de XT (ver Gram–Schmidt) e então repetindo-se tal algoritmo para encontrar a próxima componente principal. No entanto, esta estratégia simplista não é estável numericamente se mais de um pequeno número de componentes principais são exigidos, porque imprecisões nos cálculos afetarão aditivamente as estimativas de componentes principais subsequentes. Métodos mais avançados elaboram na ideia básica, como no caso do similar en:Lanczos algorithm.
Uma forma de se calcular o autovalor que corresponde a cada componente principal é medir a diferença na soma de distâncias ao quadrado entre as linhas e a média, antes e depois de subtrair-se o componente principal. O autovalor que corresponde ao componente que foi removido é igual a essa diferença.
O método NIPALS
Para dados de alta dimensionalidade, tais como os gerados nas ciências *omicas (e.g., genomica, en:metabolomics) e visão computacional, é geralmente necessário apenas calcular os primeiros componentes principais. O algoritmo en:non-linear iterative partial least squares (NIPALS) calcula t1 e p1' de X. O produto exterior, t1p1' pode então ser subtraído de X, restando a matriz residual E1. Isso pode então ser usado para calcular os componentes principais subsequentes.[14] Isso resulta numa redução dramática no tempo de cálculo já que se evita um calculo completo e explícito da matriz de covariância.
Estimação online ou sequencial
Numa situação "online" ou de "streaming", com dados chegando parte por parte em vez de serem guardados num único batch, é útil realizar uma estimativa da projeção PCA que pode ser atualizada sequêncialmente. Isso pode ser realizado eficientemente, mas exige algoritmos diferentes.[15]
Generalizações
Generalizações não lineares
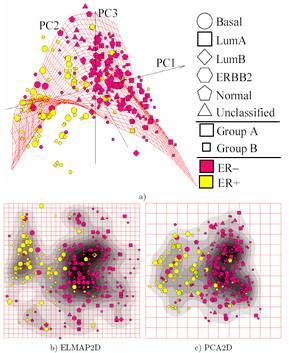
A maioria dos métodos modernos para en:nonlinear dimensionality reduction encontram suas raízes teóricas e algorítmicas no PCA ou K-means. A ideia original de Pearson era tomar uma reta (ou plano) que seja o "melhor ajuste" a um conjunto de pontos/dados. Curvas principais e variedades[18] fornecem o framework natural para a generalização do PCA e estendem a interpretação do PCA ao construirem explicitamente uma variedade para aproximação de dados, e ao codificar usando projeção matemáticas padrão sobre a variedade, como ilustrado pela Fig. Ver também o algoritmo en:elastic map e en:principal geodesic analysis.
Generalizações multilineares
No en:multilinear subspace learning, o PCA é generalizado a multilinear PCA (MPCA), o qual extrai características diretamente de representações tensoriais. MPCA é resolvido fazendo-se PCA iterativamente em cada moda do tensor. MPCA tem sido aplicado ao reconhecimento de faces, reconhecimento de andar (gait), gestos, etc. O MPCA pode ser estendido ao uncorrelated MPCA, MPCA não negativo e MPCA robusto.
Ordens mais altas
O N-way PCA pode ser realizado com modelos tais como en:Tucker decomposition, en:PARAFAC, multiple factor analysis, co-inertia analysis, STATIS, and DISTATIS.
Robustez - PCA com pesos
Apesar do PCA encontrar o método matematicamente ótimo (no sentido de minimizar o erro quadrático), ele é sensível a outliers nos dados, que produzem grandes erros que o PCA tenta evitar. Portanto, é de praxe remover os outliers ao calcular o PCA. No entanto, em alguns contextos, os outliers podem ser difíceis de se identificar de antemão. Por exemplo, em algoritmos de mineração de dados como en:correlation clustering, a atribuição de pontos a clusters e outliers não é conhecida de antemão. Uma generalização proposta recentemente de PCA[19] baseada em um PCA com pesos aumenta a robustez, associando pesos diferentes aos dados de acordo com sua relevância estimada.
Referências
- ↑ Pearson, K. (1901). «On Lines and Planes of Closest Fit to Systems of Points in Space» (PDF). Philosophical Magazine. 2 (6): 559–572
- ↑ Abdi. H.,; Williams, L.J. (2010). «Principal component analysis.». Wiley Interdisciplinary Reviews: Computational Statistics,. 2: 433-459
- ↑ Shaw P.J.A. (2003) Multivariate statistics for the Environmental Sciences, Hodder-Arnold. ISBN 0-3408-0763-6. [falta página]
- ↑ Jolliffe I.T. Principal Component Analysis, Series: Springer Series in Statistics, 2nd ed., Springer, NY, 2002, XXIX, 487 p. 28 illus. ISBN 978-0-387-95442-4
- ↑ A. A. Miranda, Y. A. Le Borgne, and G. Bontempi. New Routes from Minimal Approximation Error to Principal Components, Volume 27, Number 3 / June, 2008, Neural Processing Letters, Springer
- ↑ Fukunaga, Keinosuke (1990). Introduction to Statistical Pattern Recognition. [S.l.]: Elsevier. ISBN 0122698517
- ↑ Jonathon Shlens, A Tutorial on Principal Component Analysis. Arquivado em 15 de fevereiro de 2010, no Wayback Machine.
- ↑ Informações adicionais podem ser lidas aqui
- ↑ A.A. Miranda, Y.-A. Le Borgne, and G. Bontempi. New Routes from Minimal Approximation Error to Principal Components, Volume 27, Number 3 / June, 2008, Neural Processing Letters, Springer
- ↑ Eigenvalues function Mathematica documentation
- ↑ The Numerical Algorithms Group. «NAG Library Routine Document: nagf_mv_prin_comp (g03aaf)» (PDF). NAG Library Manual, Mark 23. Consultado em 16 de fevereiro de 2012
- ↑ The Numerical Algorithms Group. «NAG Library Routine Document: nag_mv_prin_comp (g03aac)» (PDF). NAG Library Manual, Mark 9. Consultado em 16 de fevereiro de 2012. Arquivado do original (PDF) em 24 de novembro de 2011
- ↑ Roweis, Sam. "EM Algorithms for PCA and SPCA." Advances in Neural Information Processing Systems. Ed. Michael I. Jordan, Michael J. Kearns, and Sara A. Solla The MIT Press, 1998.
- ↑ Geladi, Paul; Kowalski, Bruce (1986). «Partial Least Squares Regression:A Tutorial». Analytica Chimica Acta. 185: 1–17. doi:10.1016/0003-2670(86)80028-9
- ↑ Warmuth, M. K.; Kuzmin, D. (2008). «Randomized online PCA algorithms with regret bounds that are logarithmic in the dimension». Journal of Machine Learning Research. 9: 2287–2320
- ↑ A. N. Gorban, A. Y. Zinovyev, Principal Graphs and Manifolds, In: Handbook of Research on Machine Learning Applications and Trends: Algorithms, Methods and Techniques, Olivas E.S. et al Eds. Information Science Reference, IGI Global: Hershey, PA, USA, 2009. 28-59.
- ↑ Wang, Y., Klijn, J.G., Zhang, Y., Sieuwerts, A.M., Look, M.P., Yang, F., Talantov, D., Timmermans, M., Meijer-van Gelder, M.E., Yu, J. et al.: Gene expression profiles to predict distant metastasis of lymph-node-negative primary breast cancer. Lancet 365, 671-679 (2005); Data online
- ↑ A. Gorban, B. Kegl, D. Wunsch, A. Zinovyev (Eds.), Principal Manifolds for Data Visualisation and Dimension Reduction, LNCSE 58, Springer, Berlin – Heidelberg – New York, 2007. ISBN 978-3-540-73749-0
- ↑ Kriegel, Hans-Peter; Kröger, Peer; Schubert, Erich; Zimek, Arthur (1 de janeiro de 2008). Bertram, ed. A General Framework for Increasing the Robustness of PCA-Based Correlation Clustering Algorithms. [S.l.]: Springer Berlin Heidelberg. p. 418-435. ISBN 978-3-540-69476-2. doi:10.1007/978-3-540-69497-7_27













![{\displaystyle u[m]={1 \over N}\sum _{n=1}^{N}X[m,n]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ee0d3ca089bd8dc3a4329adc016b6664feeecded)

![{\displaystyle h[n]=1\,\qquad \qquad {\text{for }n=1,\ldots ,N}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2d8ebe795965a15f24b93098a1a74eacf7c71f01)
![{\displaystyle \mathbf {C} =\mathbb {E} \left[\mathbf {B} \otimes \mathbf {B} \right]=\mathbb {E} \left[\mathbf {B} \cdot \mathbf {B} ^{*}\right]={1 \over N}\sum _{}\mathbf {B} \cdot \mathbf {B} ^{*}](https://wikimedia.org/api/rest_v1/media/math/render/svg/20a957054eabaa333568723057f40421e6cbd92f)




![{\displaystyle D[p,q]=\lambda _{m}\qquad {\text{para }p=q=m}](https://wikimedia.org/api/rest_v1/media/math/render/svg/028e4186bb40308b570abbc0fa0e364ecd60b1e0)
![{\displaystyle D[p,q]=0\qquad {\text{for }p\neq q.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/837fc9d40f79dd7d13caa5b0f0696e931851a7f5)
![{\displaystyle g[m]=\sum _{q=1}^{m}D[q,q]\qquad \mathrm {for} \qquad m=1,\dots ,M}](https://wikimedia.org/api/rest_v1/media/math/render/svg/bfde6363a0d2c3cfa4d0310faa2ca3fc36d34aee)
![{\displaystyle W[p,q]=V[p,q]\qquad \mathrm {for} \qquad p=1,\dots ,M\qquad q=1,\dots ,L}](https://wikimedia.org/api/rest_v1/media/math/render/svg/06f15605f59da5eaa259e2a33732f58266bd478a)

![{\displaystyle {\frac {g[m=L]}{\sum _{q=1}^{M}D[q,q]}\geq 0.9\,}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2e245825877ee830b877e74c6e81869f65877163)
![{\displaystyle \mathbf {s} =\{s[m]\}=\{\sqrt {C[m,m]}\}\qquad {\text{for }m=1,\ldots ,M}](https://wikimedia.org/api/rest_v1/media/math/render/svg/8a9cbe9d7047f5093316086e86b8d6ca03a7eb93)







